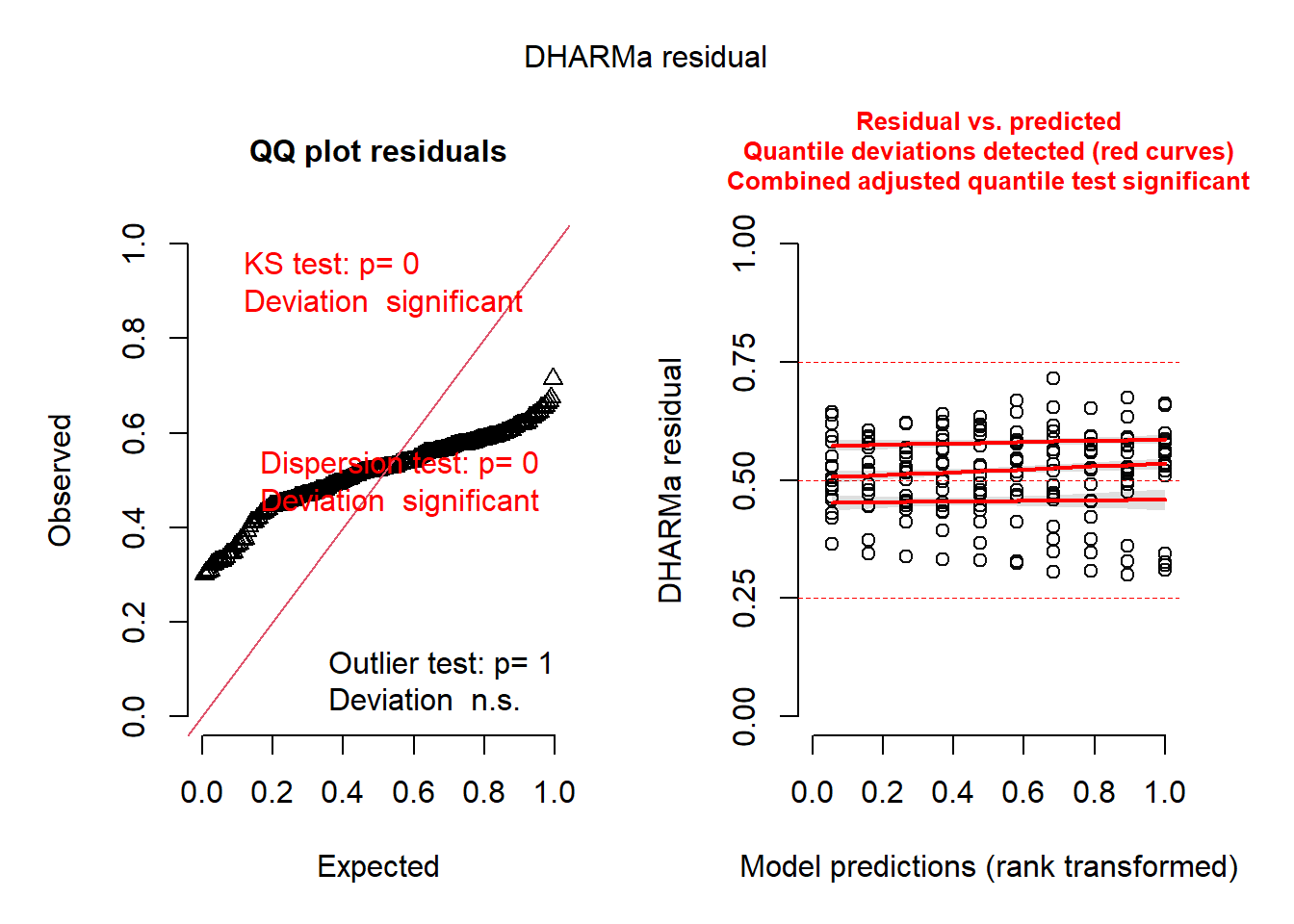
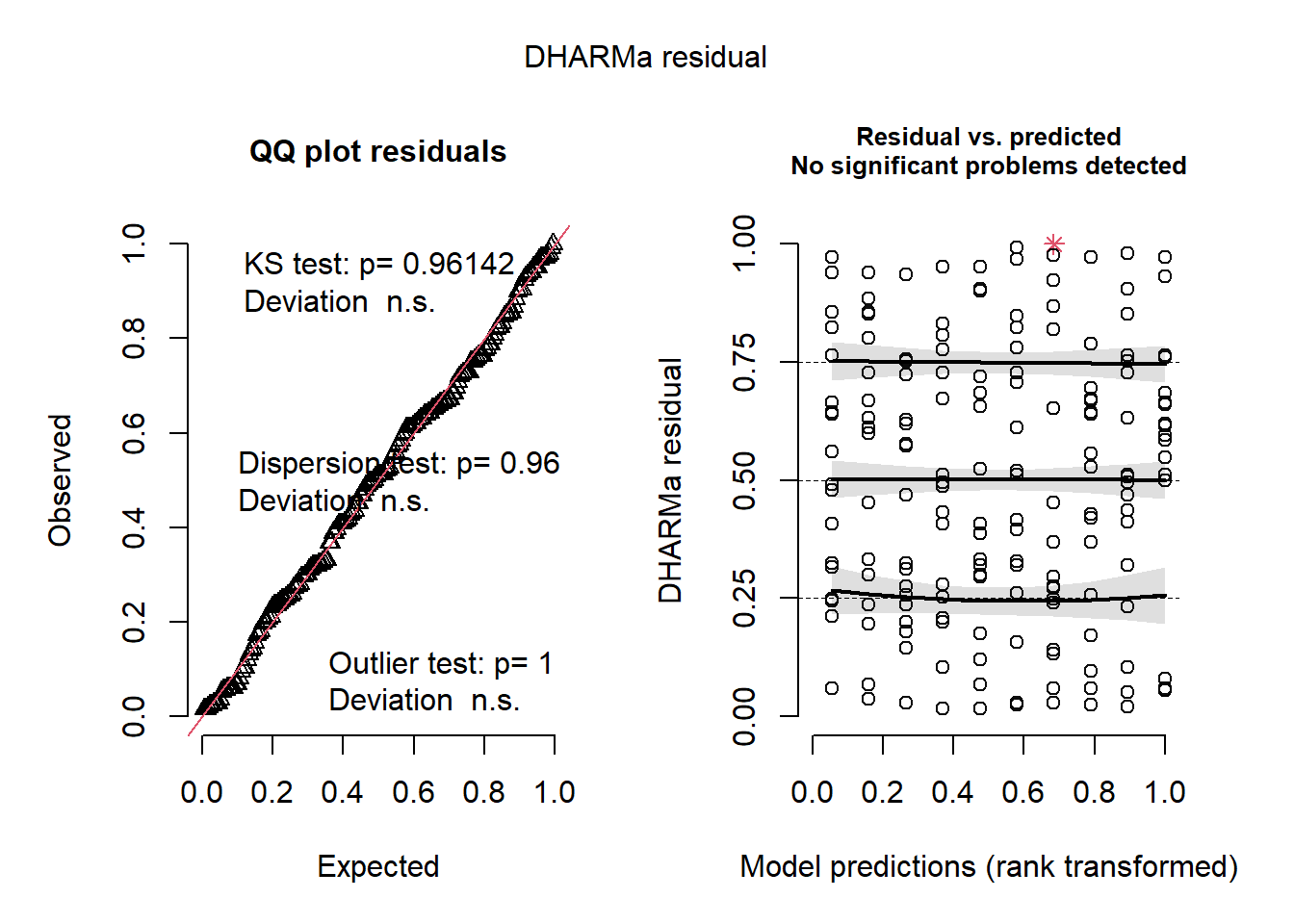
3. Interpretação
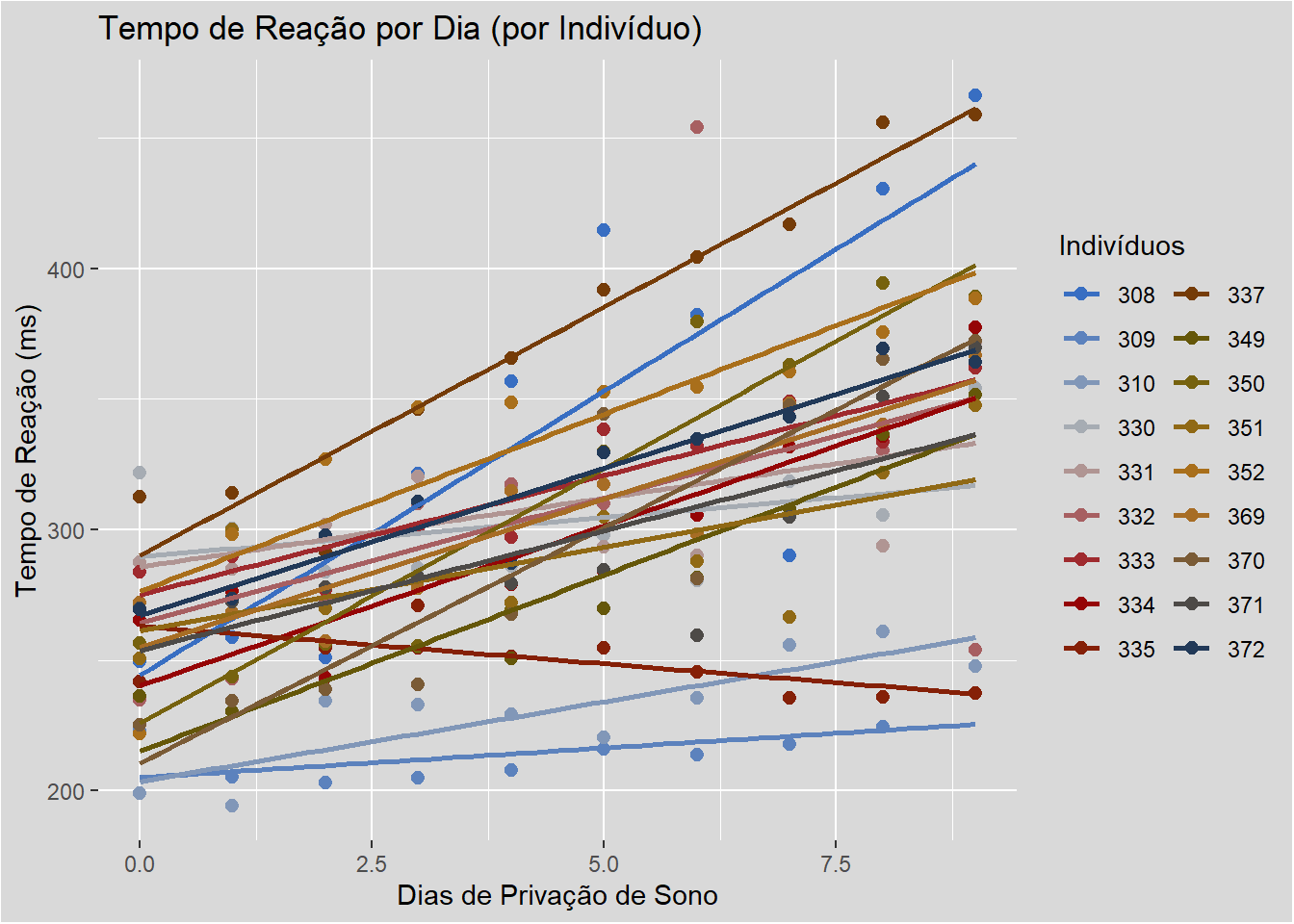
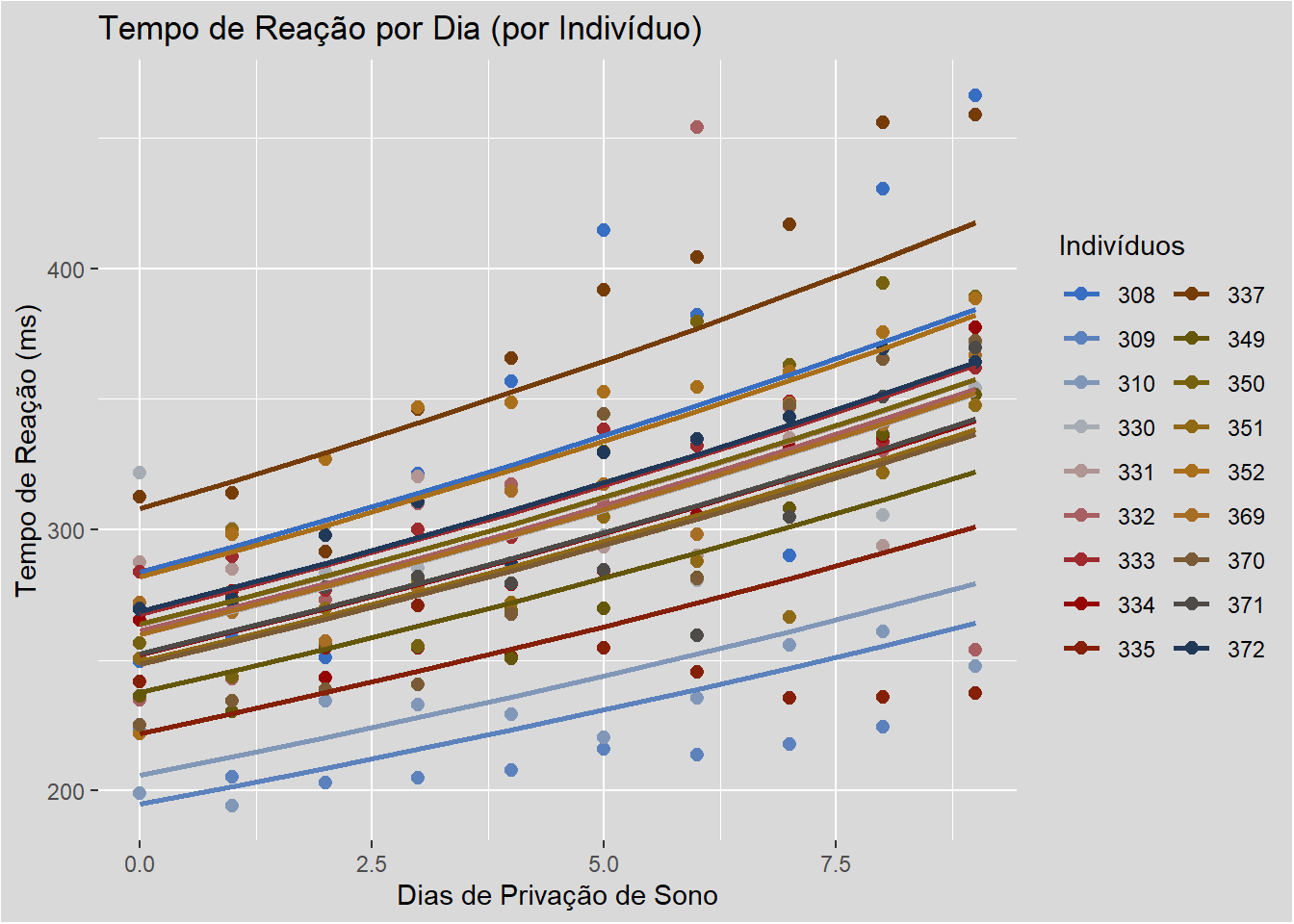
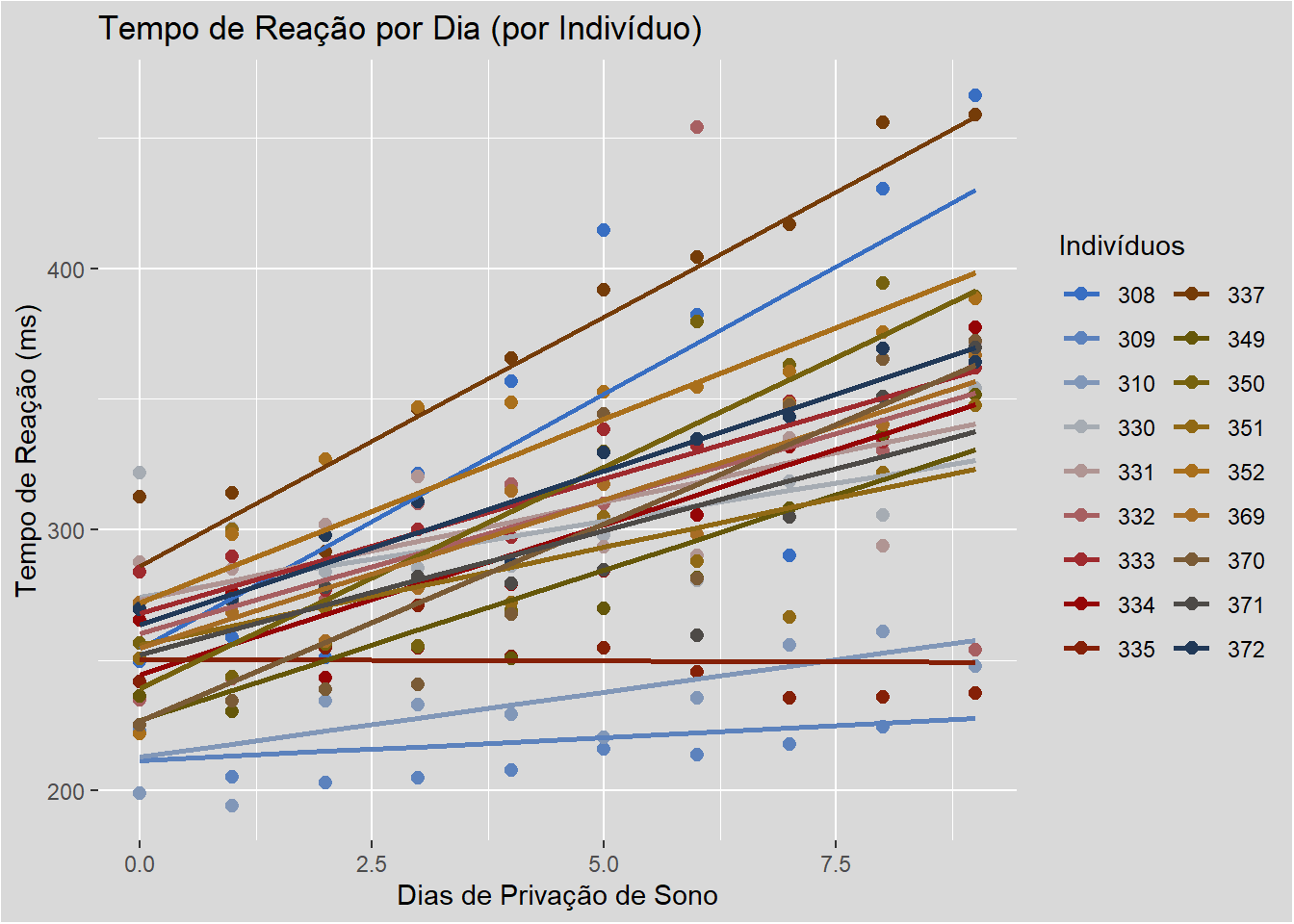
R² marginal = 0.288: Apenas 28.8% da variância em Reaction é explicada por Days (efeito fixo).
R² condicional =0.793: 79.3% da variância é explicada por Days + diferenças individuais entre Subject (efeitos aleatórios).
Quanto maior a diferença entre o R² marginal e o R² condicional, mais importante são os efeitos aleatórios no modelo.
Os efeitos aleatórios em modelos mistos (GLMMs) capturam variações não explicadas pelos efeitos fixos, mas que estão associadas a estruturas hierárquicas ou agrupamentos nos dados (ex.: indivíduos, parcelas, tempo).
A grande diferença (0.79−0.28=0.51) indica que os efeitos aleatórios dominam a explicação do modelo. Ou seja, as características individuais dos sujeitos importam mais que o efeito geral dos dias.
Se os efeitos aleatórios dominam (R² condicional muito maior que R² marginal), isso sugere que:
Portanto, se o R² condicional do seu modelo GLMM for significativamente maior que o R² marginal, isso sugere que outras variáveis não medidas — como hábitos individuais (alimentação, medicamentos, atividade física) ou características intrínsecas (genética, metabolismo) — podem estar influenciando o tempo de reação.
Isso indica que cerca de 50% da variância restante (0.79−0.28) vem de fatores não controlados, como:
Hábitos de vida (caféina, exercícios).
Uso de medicamentos (estimulantes, antidepressivos).
Condições de saúde (estresse, qualidade do sono basal).
Suponha que:
Se essas variáveis não forem medidas, seu modelo atribuirá essa variação aos efeitos aleatórios (Subject), inflando o R² condicional. Considerando isso, é necessário reconhecer as limitações das análises, pois diferenças entre indivíduos não medidas explicam parte dos resultados, e sugerir novos estudos adicionando ao modelo dados sobre hábitos de vida.